图书馆VIP生命科学与医学部刘海燕教授、陈泉教授团队开发了一种不依赖于预训练结构预测网络的蛋白质主链去噪扩散概率模型SCUBA-D,可自动从头设计主链结构,或指定功能位点生成主链结构。大量的实验结果验证了SCUBA-D的设计成功率和设计精度。相关成果以“De novo protein design with a denoising diffusion network independent of pretrained structure prediction models”为题于2024年10月9日在线发表于NatureMethods。
刘海燕教授、陈泉教授团队长期致力于发展数据驱动的蛋白质设计方法。在前期工作中,建立并实验验证了利用神经网络能量函数从头设计主链结构的SCUBA模型(Nature2022),本文报道的SCUBA-D(SCUBA-diffusion)模型,是基于深度学习的主链设计算法的迭代升级。SCUBA-D能够基于不同输入执行多类蛋白质结构设计任务(图a)。在模型设计上,通过在扩散模型训练中引入对抗损失(adversarial loss)(图b),避免生成模型产生物理上不可行的结构,实现了高成功率的主链结构设计。由于没有使用已有结构预测网络作为预训练降噪网络,SCUBA-D能够在设计中避免对已知天然结构的过度偏好,可发现已有模型在可设计蛋白质结构空间中的盲区。
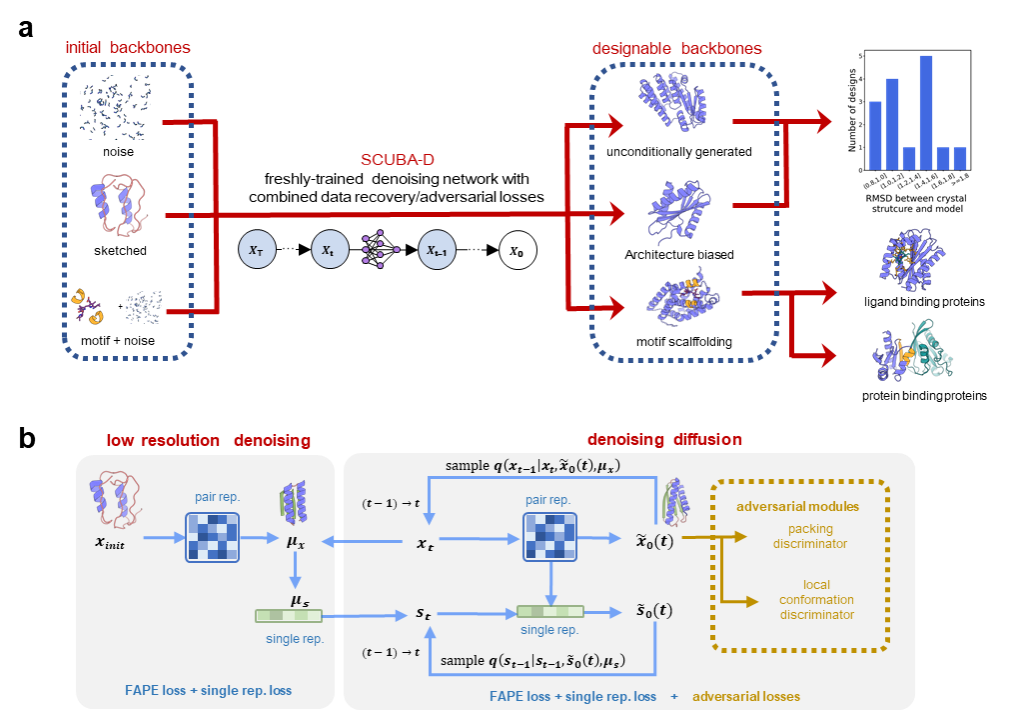
图. 用SCUBA-D模型进行蛋白质主链设计的原理。(a)SCUBA-D可基于噪声或者用户定义的结构草图设计新主链结构,也可以基于给定的含功能位点的局部结构设计新的完整主链。(b)包含对抗损失函数的SCUBA-D模型架构。
团队对SCUBA-D在多类蛋白质从头设计任务中的应用进行了实验验证。针对单体结构从头设计任务,团队对共计70条设计序列进行了实验表征,其中近80%的序列(53条)可溶表达,实验解析的16个高分辨晶体结构与目标结构高度一致(主链原子位置均方根位移在0.96到2.11 Å之间)。在小分子结合蛋白设计任务中,团队对非经典血红素降解酶进行了保留结合位点的主链结构重设计,对设计的12条序列进行实验验证,其中5条具有与血红素的结合能力,三条序列与血红素的亲和力与天然蛋白相当或高于天然蛋白。在结合蛋白设计任务中,30个人工设计的Ras结合蛋白14个与Ras有相互作用,其中3个设计蛋白与Ras的结合亲和力与天然蛋白相当,复合物晶体结构更进一步验证了设计的精确度。
中国科大生命科学与医学部刘海燕教授、陈泉教授为该论文通讯作者;生命科学与医学部博士生刘宇枫、王晟为该论文共同第一作者,论文合作者包括来自科大讯飞和元构生物等企业的研究人员。该研究工作得到了科技部、国家自然科学基金委、中国科学院等多项基金资助支持。
原文链接:https://www.nature.com/articles/s41592-024-02437-w
(生命科学与医学部、微尺度物质科学国家研究中心、细胞动力学教育部重点实验室、科研部)